Inferência Causal
Rerandomização e ajuste de regressão
ctrucios@unicamp.br
Instituto de Matemática, Estatística e Computação Científica (IMECC),
Universidade Estadual de Campinas (UNICAMP).
Introdução
Introdução
- Experimentos randomizados são o padrão ouro para estimar efeitos causais, porque a randomização equilibra, em média, todos os possíveis fatores de confusão (a.k.a. covariáveis). Contudo, pode acontecer que, em um determiado experimento, CRE crie grupos que são desbalanceados em alguma covariável importante.
- Esse desbalanceamento traz consequências (já discutidas na aula anterior).
- Nestes casos, continuamos com o experimento como esta ou re-randomizamos e continuamos o experimento com grupos balanceados?
Introdução
- Estratificação e pós-estratificação são duais para covariável discreta no planejamento e análise de experimentos randomizados.
- Mas como lidar com covariáveis multidimensionais e/ou contínuas?
- Categorizar poderia ser uma respostas mas é longe de ser uma solução ótima 😿.
- Rerandomizar e ajuste de regressão são o caminho a seguir 😄.
| Planejamento | Análise | |
|---|---|---|
| Covariável Discreta | Estratificação | Pós-estratificação |
| Covariável Geral | Rerandomizar | Ajuste de regressão |
Rerandomizar
Rerandomizar
- Considere uma polulação com \(n\) unidades experimentais, das quais \(n_1\) recebem o tratamento e \(n_0\) o controle e seja \(\textbf{Z} = (Z_1, \cdots, Z_n)\) o vetor indicador de tratamento.
- Para cada \(i\), seja \(X_i \in \mathbb{R}^K\) o vetor de covariáveis (discretas ou contínuas).
- Denote, \(\textbf{X}_{n \times K} = (X_1, \cdots, X_n)'\) e por simplicidade assumiremos que \(n^{-1} \displaystyle \sum_{i = 1}^n X_i = 0.\)
Sob CRE, a diferença de médias das covariáveis, \[\hat{\tau}_X = n_1^{-1} \displaystyle \sum_{i = 1}^n Z_i X_i - n_0^{-1}\sum_{i = 1}^n (1- Z_i) X_i,\] tem \(\mathbb{E}(\hat{\tau}_X) = 0\) e \(\mathbb{V}(\hat{\tau}_X) = \dfrac{n}{n_1 n_0}S^2_X\), em que \(S^2_X = (n - 1)^{-1} \displaystyle \sum_{i = 1}^nX_i X_i'.\)
Rerandomizar
- Randomizar, em média, equilibra as covariáveis.
- Contudo, na alocação tratamento-controle realizada, é possível que covariáveis ainda sejam desbalanceadas, comprometendo a validade dos resultados.
- Para evitar este desbalancemento, rerandomizamos.
- Rerandomizar consite em refazer a randomização até que algum critério pre-especificado de equilibrio das covariáves seja atingido.
- Este procedimento é computacionalmente caro (comparado com estratificar, por exemplo), mas hoje em dia isto não é mais um problema.
Rerandomizar
Uma forma de avaliar a diferença entre os grupos de tratamento e controle é através da distância de Mahalanobis:
\[M = \hat{\tau}_X' \Big (\underbrace{\dfrac{n}{n_1n_0}S^2_X}_{\mathbb{V}(\hat{\tau}_X)} \Big )^{-1}\hat{\tau}_X.\]
Lema
\(M\) permanece o mesmo se transformamos \(X_i\) em \(b_0 + B X_i\) para todas as unidades \(i = 1, \cdots, n\), em que \(b_0 \in \mathbb{R}^K\) e \(B \in \mathbb{R}^{K \times K}.\)
Sob CRE e para valores grandes de \(n\), \(M \sim \chi^2_K.\) Assim, \(M\) pode ter valores realizados grandes, afinal, \(M\) tem média \(K\) e variância \(2K\).
Rerandomizar
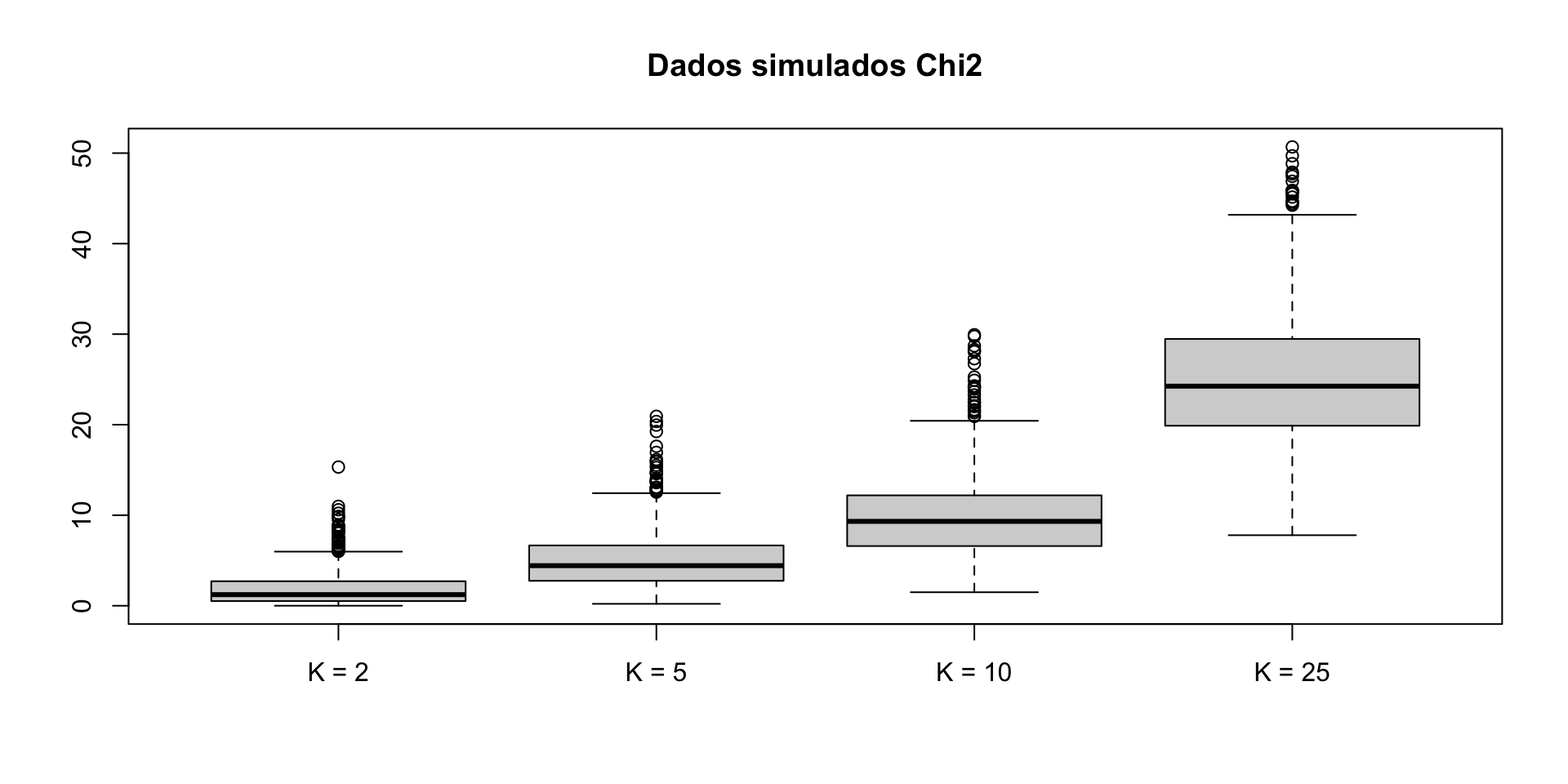
Rerandomizar
Rerandomizar
- Rerandomizar evita o desbalanceamento das covariáveis descartando as alocações de tratamento (\(\textbf{z}\)) nas quais \(M\) é grande.
Definição (ReM): Rerandomizaçao utilizando a distância de Mahalanobis (\(M\))
Selecione \(\textbf{Z}\) de um CRE e aceite este sss \[M \leq a,\] para algum valor predeterminado de \(a > 0.\)
Como escolher a?
Uma prática comúm é escolher \(a\) pequeno ou utilizar algum quantil da \(\chi^2_K\).
Rerandomizar
- Uma outra forma de escolher \(a\) é fixando primeiro \(p_a\) tal que \[p_a = P(M \leq a),\] em que \(p_a\) é a proporção de todas as randomizações consideradas aceitáveis pelo critério de rerandomização.
- Na prática, a escolha de \(p_a\) é feita em termos do PRIV.
Percent Reduction in Variance (PRIV)
\[PRIV = 100 \times \Big (\dfrac{\mathbb{V}(\hat{\tau}_X) - \mathbb{V}_a(\hat{\tau}_X)}{\mathbb{V}(\hat{\tau}_X)} \Big) = 100 \times \Big ( 1 - \dfrac{P(\chi_{k+2}^2 \leq a)}{P(\chi_{k}^2 \leq a)} \Big)\]
Rerandomizar
Exemplo
- \(p_a = 0.001 \rightarrow a = 1.478743 \rightarrow PRIV \approx 88\%\)
- \(p_a = 0.0001 \rightarrow a = 0.8889204 \rightarrow PRIV \approx 93\%\)
Note que, o número de rerandomizações até o primeiro aceitável é \(1/p_a\). Dependendo do tempo computacional necessário (o que tem testes de permutação é um fator importante), o 5% adicional na redução da variância pode ou não valer a pena.
Rerandomizar
Rerandomizar
Como analisar dados sob ReM?
- Sempre podemos utilizar FRT, desde que simulemos \(\textbf{Z}\) sob a restrição de que \(M \leq a\) (Morgan e Rubin, 2012).
- Não temos propriedades em amostras finitas sem assumir a hipótese nula forte, mas temos alguns resultados assintóticos que nos ajudarão a testar a hipótese nula fraca.
Notação
Seja \(L_{K,a} \sim D_1 | \textbf{D}'\textbf{D} \leq a,\) em que \(\textbf{D} = (D_1, \cdots, D_K) \sim N_K(0, I)\) e seja \(\varepsilon \sim N(0,1)\), então \(L_{K, a}\) e \(\varepsilon\) são independentes
Rerandomizar
Teorema
Sob ReM com \(M \leq a\), quando \(n \rightarrow \infty\) e se as condições (1)–(3) acontecem, então \[\hat{\tau} - \tau \sim \sqrt{\mathbb{V}(\hat{\tau})} \Big \{\sqrt{R^2}L_{K,a} + \sqrt{1 - R^2}\varepsilon \Big \},\] em que \(\mathbb{V}(\hat{\tau}) = \dfrac{S^2(1)}{n_1} + \dfrac{S^2(0)}{n_0} - \dfrac{S^2(\tau)}{n}\) e \(R^2 = \mathbb{C}or^2(\hat{\tau}, \hat{\tau}_X)\) (sob CRE).
Condições:
- \(n_1/n \rightarrow k_1\) e \(n_1/n \rightarrow k_2\) com \(0 \leq k_1, k_2 < \infty\).
- \(\{X_i, Y_i(1), Y_i(0), \tau_i \}\) tem variância finita.
- \(\max_{1 \leq i \leq n} \{Y_i(1)-\bar{Y}(1) \}^2 / n \rightarrow 0\), \(\max_{1 \leq i \leq n} \{Y_i(0)-\bar{Y}(0) \}^2 / n \rightarrow 0\) e \(\max_{1 \leq i \leq n} X_i'X_i / n \rightarrow 0\).
- Quando \(a = \infty\), \(\hat{\tau}- \tau \sim \sqrt{\mathbb{V}(\hat{\tau})}\varepsilon\).
- Quando \(a \rightarrow 0\), \(\hat{\tau}- \tau \sim \sqrt{\mathbb{V}(\hat{\tau})(1 - R^2)}\varepsilon\).
Rerandomizar
\[R^2 = \mathbb{C}or(\hat{\tau}, \hat{\tau}_X) = \dfrac{n_1^{-1}S^2(1|X) + n_0^{-1}S^2(0|X) - n^{-1}S^2(\tau | X)}{_1^{-1}S^2(1) + n_0^{-1}S^2(0) - n^{-1}S^2(\tau)},\] em que \(S^2(1)\), \(S^2(0)\), \(S^2(\tau)\) são as variâncias de \(\{Y_i(1)\}_{i = 1}^n\), \(\{Y_i(0)\}_{i = 1}^n\), \(\{\tau_i\}_{i = 1}^n\) e \(S^2(1|X)\), \(S^2(0|X)\), \(S^2(\tau|X)\) são as variâncias das suas projeções em \((1, X)\).
Quando \(0<a<\infty\), a distribuição de \(\hat{\tau}\) é bem mais complexa mas é mais concentrada em \(\tau\). Assim, \(\hat{\tau}\) é mais preciso sob ReM do que sob CRE.
Observação: Se ignorarmos o ReM e ainda utilizarmos o intervalo de confiança baseado na fórmula de variância de Neyman (1923) e na aproximação Normal, este será excessivamente conservador.
Ajuste de regressão
Ajuste de regressão

Obviamente não somos Marty McFly e não podemos voltar no tempo para utilizar rerandomização. Contudo, não todo está perdido e é aqui que ajuste de regressão entra no jogo.
Ajuste de regressão
FRT ajustado por covariável
As covariáveis \(\textbf{X}\) são todas fixas e, sob \(H_{0F}\), os resultados observados também são fixos. Assim, podemos simular a distribuição de qualquer test \(T = T(\textbf{Y}, \textbf{Z}, \textbf{X})\) e calcular p-valores. A ideia central do FRT continua a mesma na presença de covariáveis.
Existem duas estratégias gerais para construir a estatística de teste, as quais são apresentadas a seguir:
Ajuste de regressão
Definição: estratégia de pseudo-resultado
Podemos construir a estatística de teste baseados nos residuos (\(\hat{\varepsilon}\)) do modelo ajustado (considerando \(\textbf{Y}\) como variável dependente e \(\textbf{X}\) como independentes) e utilizar os resíduos como o pseudo-resultado para construir o teste.
Definição: estratégia da “saida” do modelo (model-output)
Podemos ajustar o modelo (considerando \(\textbf{Y}\) como variável dependente e \(\textbf{X}\) e \(\textbf{Z}\) como independentes) para obter o coeficiente de \(\textbf{Z}\) e utilizá-lo como estatística de teste.
Na primeira estratégia, precisamos rodar o modelo de regressão apenas uma vez. Já na segunda estratégia precisamos rodar o modelo várias vezes.
Observação: Um dos modelos ajustados mais utilizado é regressão linear (por MQO), mas nada impede que outras abordagens tais como regressão logística ou mesmo modelos de machine learning sejam utilizados.
Ajuste de regressão
E se não quisermos testar \(H_{0F}\) mas \(H_{0N}\)?
Os seguintes métodos focam na estimação do efeito médio causal, \(\tau\).
ANCOVA
- Fisher (1925) propõe ANCOVA para melhorar a eficiência.
- A ideia básica é ajustar um modelo de regressão por MQO, \[y_i = \beta_0 + \beta_1 z_i + \beta_2 x_{i1} + \cdots, \beta_{k+1} x_{ik} + u,\] e considerar \(\hat{\beta}_1\) como um estimador para \(\tau\), que será denotado aqui por \(\hat{\tau}_F\).
Ajuste de regressão
Freedman
David Freedman (2008) reanalisou ANCOVA sob o olhar dos resultados potenciais e encontrou o seguinte:
- \(\hat{\tau}_F\) é viesado mas a simple diferença de médias, \(\hat{\tau}\), é não viesado.
- A variância assintótica de \(\hat{\tau}_F\) pode ser ainda maior do que a de \(\hat{\tau}\) quando \(n_1 \neq n_0\).
- O erro padrão do MQO é inconsistente para o verdadeiro erro padrão de \(\hat{\tau}_F\) sob CRE.
Lin
Winston Lin na sua tese de doutorado (2013), respondeu a algumas das críticas do Freedman e encontrou o seguinte:
- O vies de \(\hat{\tau}_F\) é pequeno em grandes amostras e quando \(n \rightarrow \infty\), o vies \(\rightarrow 0\).
- A eficiência assintótica de \(\hat{\tau}_F\) pode ser melhorada utilizando o coeficiente de \(Z_i\) resultante da regressão de \(\textbf{Y}\) sob \((1, \textbf{Z}, \textbf{X}, \textbf{Z} \times \textbf{X})\), denotaremos o estimador por \(\hat{\tau}_L\). Além disso, o erro padrão EHW é um estimador conservador do verdadeiro erro padrão de \(\hat{\tau}_L\) sob CRE.
- O erro padrão EHW para \(\hat{\tau}_F\) ( regressão de \(\textbf{Y}\) sob \((1, \textbf{Z}, \textbf{X})\)) é um estimador conservador para o verdadeiro erro padrão de \(\hat{\tau}_F\)
Lin e SRE
Lin e SRE
Pode ser o caso de termos um SRE, estratificado numa variável discreta \(C\) e também termos outras covariáveis \(X\). Se todos os estratos são grandes, podemos obter um estimador de Lin como \[\hat{\tau}_{L,S} = \displaystyle \sum_{k = 1}^K \pi_{[k]} \hat{\tau}_{L,[k]},\] e um estimador (conservador) de \(\mathbb{V}(\hat{\tau}_{L,S})\) dado por \[\hat{V}_{L,S} = \displaystyle \sum_{k = 1}^K \pi_{[k]}^2 \hat{V}_{EHW, [k]}\]
Exemplo
Exemplo
[Angrist et al. (2009)]
- Os dados vem de um experimento avaliando diferentes estratégias para melhorar o desempenho acadêmico nos calouros em uma universidade do Canada.
- Por simplicidade, vamos focar apenas no grupo controle (
control) e no tratamento que consiste em serviços de apoio acadêmico e incentivos financeiros (sfsp). - O resultado de interesse é o CR no final do primero ano (
GPA_year1). - Dados faltantes são imputados com a média.
- As covariáveis são genero (
gender) e nota do vestibular (gpa0) - Os dados estão disponíveis aqui
Exemplo
[Angrist et al. (2009)]
Code
library(foreign)
library(dplyr)
library(car)
dados <- read.dta("datasets/star.dta")
dados <- dados |>
filter(control == 1 | sfsp == 1) |>
mutate(GPA_year1 = ifelse(is.na(GPA_year1), mean(GPA_year1, na.rm = TRUE), GPA_year1)) |>
select(GPA_year1, sfsp, female, gpa0)
y <- dados$GPA_year1
z <- dados$sfsp
x <- dados[, c("female", "gpa0")]
# Unadjusted estimator (Neyman)
modelo_unadj <- lm(y ~ z)
tau_unadj <- coef(modelo_unadj)[2]
se_unadj <- sqrt(hccm(modelo_unadj, type = "hc2")[2, 2])
c(tau_unadj, se_unadj) z
0.05182942 0.07753077 Exemplo
Exemplo
Simulação
Simulação
Code
library(ggplot2)
x2 <- cbind(x, x[, 2]^2)
dados_sim <- data.frame(y = y, z = z, x2 = x2)
y1lm <- lm(y ~ x2, weights = z, data = dados_sim)
sigma1 <- summary(y1lm)$sigma
y0lm <- lm(y ~ x2, weights = 1 - z, data = dados_sim)
sigma0 <- summary(y0lm)$sigma
a <- 0.05
mc <- 2000
n1 <- sum(z)
n0 <- sum(1 - z)
n <- n1 + n0
rescale <- 0.2
y1_sim <- predict(y1lm, data = dados_sim) + rnorm(n)*sigma1*rescale
y0_sim <- predict(y0lm, data = dados_sim) + rnorm(n)*sigma0*rescale
tau <- mean(y1_sim - y0_sim)
tau_hat_cre <- rep(0, mc)
tau_hat_lim <- rep(0, mc)
tau_hat_rem <- rep(0, mc)
tau_hat_rem_lim <- rep(0, mc)
for (i in 1:mc) {
## CRE
z_cre <- sample(z)
y_cre <- z_cre * y1_sim + (1 - z_cre) * y0_sim
tau_hat_cre[i] <- mean(y_cre[z_cre == 1]) - mean(y_cre[z_cre == 0])
## Regression Adjustement (Lim)
tau_hat_lim[i] <- lm(y_cre ~ z_cre * x)$coef[2]
## ReM
z_rem <- rem(x, n1, n0, a)
y_rem <- z_rem * y1_sim + (1 - z_rem) * y0_sim
tau_hat_rem[i] <- mean(y_rem[z_rem == 1]) - mean(y_rem[z_rem == 0])
tau_hat_rem_lim[i] = lm(y_rem ~ z_rem * x)$coef[2]
}Simulação
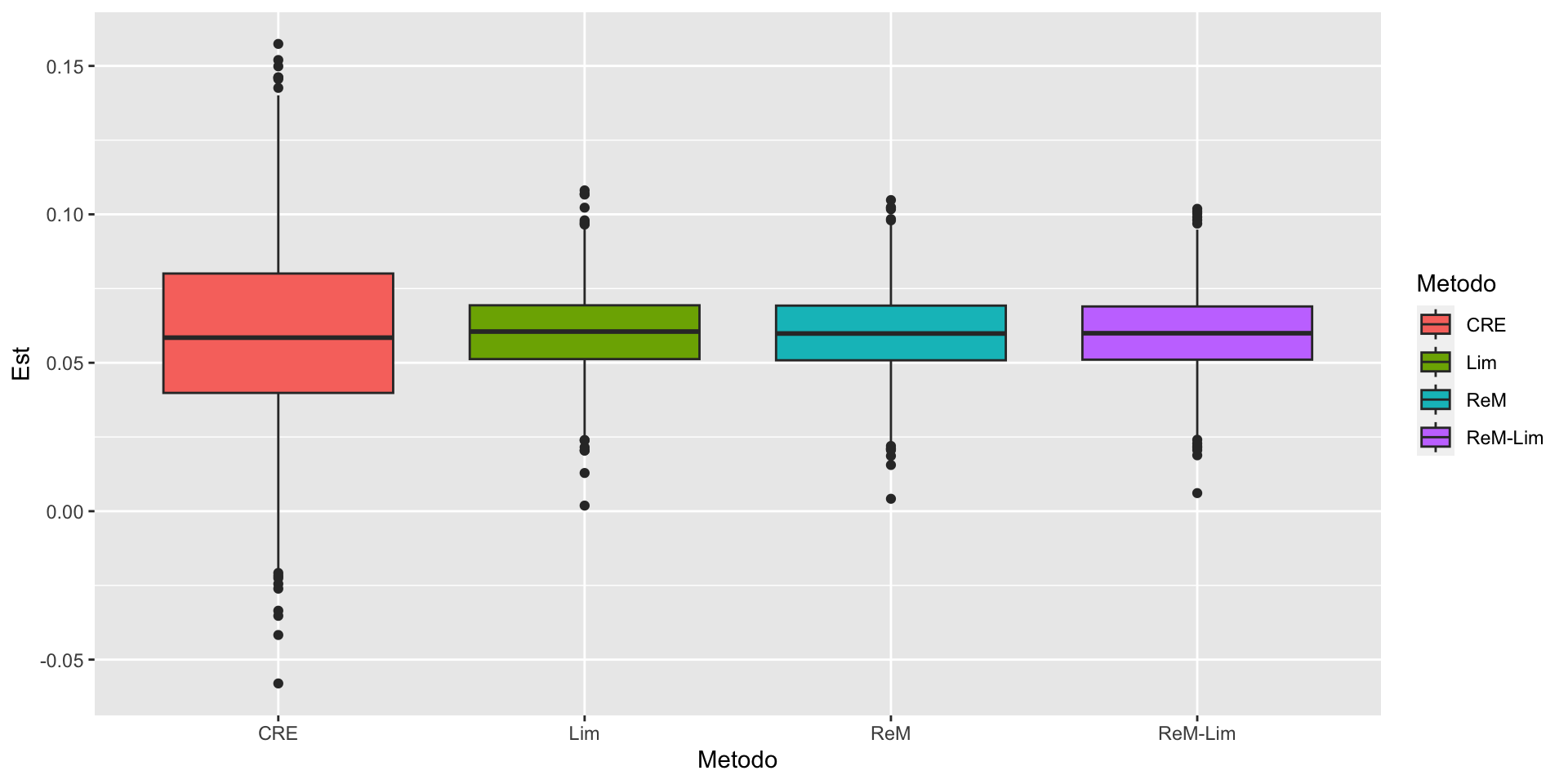
Simulação
Observação:
No experimento de simulação temos, além dos métodos vistos na aula de hoje, utilizado o estimador de Lim num contexto de rerandomização. De fato, Li e Ding (2020), mostram que é possível fazer isto e que, em geral, está combinação melhora o equilibrio da covariável tanto na parte do desenho amostral quando na parte de análise, dando uma maior eficiencia.
Importante
Li et al. (2018) mostram quem, quando \(a\) é pequeno, a distribuição assintótica de \(\hat{\tau}\) sob ReM e a distribuição assintócica de \(\hat{\tau}_L\) sob CRE são quase idênticas.
Referências
- Peng Ding (2023). A First Course in Causal Inference. Capítulo 6.
- Morgan, K. L., & Rubin, D. B. (2012). Rerandomization to improve covariate balance in experiments.
- Angrist, J., Lang, D., & Oreopoulos, P. (2009). Incentives and services for college achievement: Evidence from a randomized trial. American Economic Journal: Applied Economics, 1(1), 136-163.
- Li, X., & Ding, P. (2020). Rerandomization and regression adjustment. Journal of the Royal Statistical Society Series B: Statistical Methodology, 82(1), 241-268.
- Li, X., Ding, P., & Rubin, D. B. (2018). Asymptotic theory of rerandomization in treatment–control experiments. Proceedings of the National Academy of Sciences, 115(37), 9157-9162.

Carlos Trucíos (IMECC/UNICAMP) | ME920/MI628 - Inferência Causal | ctruciosm.github.io